Kmeans
算法步骤
- 从样本中选择 K 个点作为初始质心(完全随机)
- 计算每个样本到各个质心的距离,将样本划分到距离最近的质心所对应的簇中
- 计算每个簇内所有样本的均值,并使用该均值更新簇的质心
- 重复步骤 2 与 3 ,直到达到以下条件之一:
- 质心的位置变化小于指定的阈值(默认为 0.0001)
- 达到最大迭代次数
欧氏距离
衡量的是多维空间中两个点之间的绝对距离
在二维和三维空间中的欧氏距离就是两点之间的实际距离
计算方法:对应坐标值相减的平方和再开方
$$ dist(X,Y)=\sqrt{\sum_{i=1}^{n}(x_i-y_i)^2} $$对于二维来说,就是
$$ dist(X,Y)=\sqrt{(x_1-y_1)^2+(x_2-y_2)^2} $$注意理解输入?输出?类别中心?迭代过程中做什么?
编程实现
| |
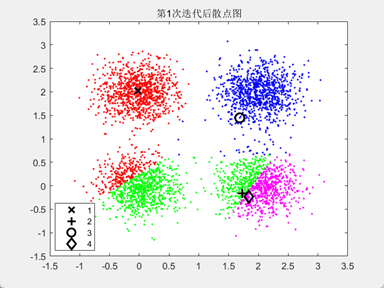
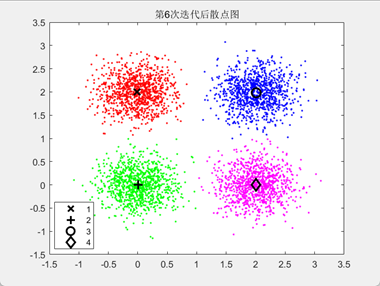
缺点
容易受初始质心的影响;算法聚类时,容易产生空簇;算法可能收敛到局部最小值。
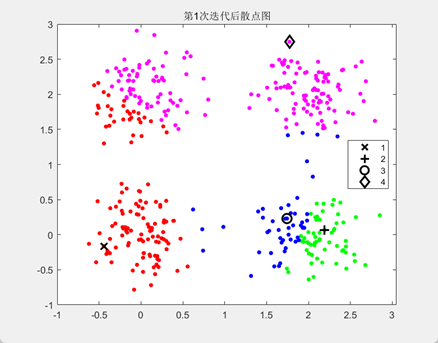
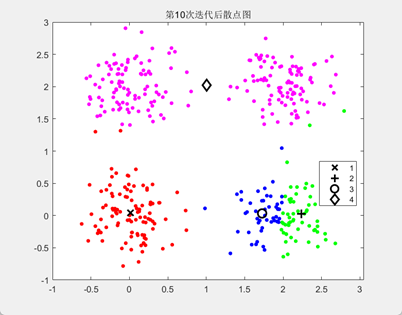
解决办法:重新执行K-means算法,重新分配质心
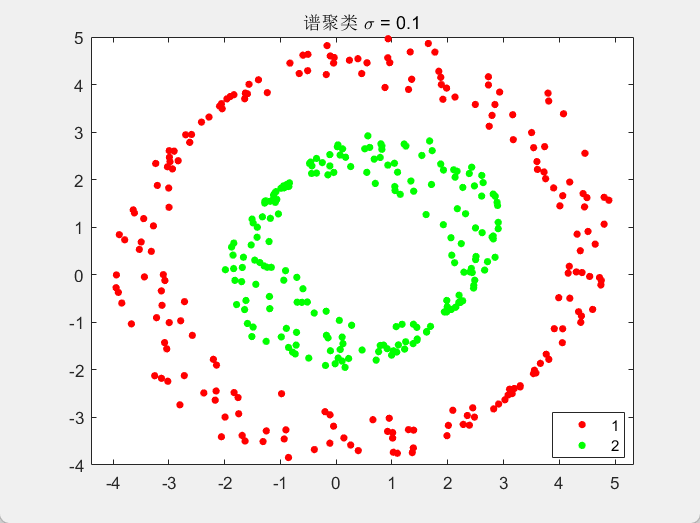
谱聚类(Spectral Clustering)
算法思想
把所有的数据看做空间中的点,这些点之间可以用边连接起来。距离较远的两个点之间的边权重值较低,而距离较近的两个点之间的边权重值较高,通过对所有数据点组成的图进行切图,让切图后不同的子图间边权重和尽可能的低,而子图内的边权重和尽可能的高,从而达到聚类的目的。
度矩阵D
对于一个图$G$,我们一般用点的集合$V$和边的集合$E$来描述。即为$G(V,E)$。其中$V$即为我们数据集里面所有的点$(v_1, v_2,…v_n)$。对于$V$中的任意两个点,可以有边连接,也可以没有边连接。我们定义权重$w_{ij}$为点$v_i$和点$v_j$之间的权重。由于我们是无向图,所以$w_{ij} = w_{ji}$。
对于有边连接的两个点$v_i$和$v_j$,$w_{ij} > 0$,对于没有边连接的两个点$v_i$和$v_j$,$w_{ij} = 0$。对于图中的任意一个点$v_i$,它的度$d_i$定义为和它相连的所有边的权重之和,即
$$ d_i = \sum\limits_{j=1}^{n}w_{ij} $$利用每个点度的定义,我们可以得到一个nxn的度矩阵$D$,它是一个对角矩阵,只有主对角线有值,对应第i行的第i个点的度数,定义如下:
$$ \mathbf{D} = \left( \begin{array}{ccc} d_1 & \ldots & \ldots \\\\ \ldots & d_2 & \ldots \\\\ \vdots & \vdots & \ddots \\\\ \ldots & \ldots & d_n \end{array} \right) $$利用所有点之间的权重值,我们可以得到图的邻接矩阵$W$,它也是一个nxn的矩阵,第i行的第j个值对应我们的权重$w_{ij}$。
除此之外,对于点集$V$的的一个子集$A \subset V$,我们定义:
$$ |A|: = 子集A中点的个数 $$$$ vol(A): = \sum\limits_{i \in A}d_i $$相似矩阵
构建邻接矩阵$W$的方法有三类:
$\epsilon$-邻近法
它设置了一个距离阈值$\epsilon$,然后用欧式距离$s_{ij}$度量任意两点$x_i$和$x_j$的距离。 然后根据$s_{ij}$和$\epsilon$的大小关系,来定义邻接矩阵$W$。距离小于$\epsilon$为$\epsilon$,距离大于$\epsilon$为0
K邻近法
利用KNN算法遍历所有的样本点,取每个样本最近的k个点作为近邻,只有和样本距离最近的k个点之间的$w_{ij} > 0$。但是这种方法会造成重构之后的邻接矩阵W非对称。
全连接法(最常用)
相比前两种方法,第三种方法所有的点之间的权重值都大于0,因此称之为全连接法。可以选择不同的核函数来定义边权重,常用的有多项式核函数,高斯核函数和Sigmoid核函数,最常用的是高斯核函数RBF。
💡 exp(x)表示$e^x$
拉普拉斯矩阵
拉普拉斯矩阵$L= D-W$
$D$即为度矩阵,它是一个对角矩阵。$W$为邻接矩阵。
无向图切图
对于无向图$G$的切图,我们的目标是将图$G(V,E)$切成相互没有连接的k个子图,每个子图点的集合为:$A_1,A_2,..A_k$,它们满足$A_i \cap A_j = \emptyset$,且$A_1 \cup A_2 \cup … \cup A_k = V$.
对于任意两个子图点的集合$A, B \subset V$, $A \cap B = \emptyset$, 我们定义A和B之间的切图权重为:
$$ W(A, B) = \sum\limits_{i \in A, j \in B}w_{ij} $$最小化切图
$$ \min cut(A,B)=\sum\limits_{i \in A, j \in B}w_{ij} $$RatioCut切图
$$ RatioCut(A,B)=cut(A,B)(\frac{1}{|A|}+\frac{1}{|B|}) $$Ncut切图
$$ Ncut(A,B)=cut(A,B)(\frac{1}{vol(A)}+\frac{1}{vol(B)}) $$
算法流程
最常用的相似矩阵的生成方式是基于高斯核距离的全连接方式,最常用的切图方式是Ncut。而到最后常用的聚类方法为K-Means。
输入:样本集D=$(x_1,x_2,…,x_n)$,相似矩阵的生成方式, 降维后的维度$k_1$, 聚类方法,聚类后的维度$k_2$
输出: 簇划分$C(c_1,c_2,…c_{k_2})$.
- 根据输入的相似矩阵的生成方式构建样本的相似矩阵S
- 根据相似矩阵S构建邻接矩阵W,构建度矩阵D
- 计算出拉普拉斯矩阵L
- 构建标准化后的拉普拉斯矩阵$D^{-1/2}LD^{-1/2}$
- 计算$D^{-1/2}LD^{-1/2}$最小的$k_1$个特征值所各自对应的特征向量$f$
- 将各自对应的特征向量$f$组成的矩阵按行标准化,最终组成$n \times k_1$维的特征矩阵F
- 对F中的每一行作为一个$k_1$维的样本,共n个样本,用输入的聚类方法进行聚类,聚类维数为$k_2$
- 得到簇划分$C(c_1,c_2,…c_{k_2})$
编程实现
| |
简化考试专用代码
| |
